Salud Animal
Influenza Aviar, el cambio es lo único constante
Para leer más contenidos de aviNews LATAM 2025 Marzo
Salud Animal
Para leer más contenidos de aviNews LATAM 2025 Marzo
Historia de la Influenza Aviar
El virus de la Influenza Aviar fue descrito por primera vez en 1878 en Italia, por Edoardo Bellarmino Perroncito, quien la describió como una enfermedad contagiosa en las aves, con alta mortalidad.
En 1880 Divolta y Delprato describieron las diferencias con el cólera aviar y llamaron a esta enfermedad Typhus exudiatious gallinarum.
El primer registro de Influenza Aviar en Norteamerica es en 1924 en la Ciudad de Nueva York, donde se presentaron pérdidas importantes en los mercados de aves vivas.
A partir de ese momento la enfermedad empezó a diseminarse por otros Estados, como Connecticut, Missouri e Indiana, en todos los casos asociados a la importación de aves de áreas donde previamente se había presentado la enfermedad.
En 1925 se tomaron medidas de cuarentena, despoblación y desinfección, con lo que se logró erradicar la enfermedad antes de que se dispersara por todo Estados Unidos.
El siguiente brote del que se tiene reporte es en 1983 y se trata de un virus H5N2, este subtipo continúa en circulación y los virus de Influenza Aviar actualmente continúan siendo un problema importante en la avicultura mundial.
Los virus influenza pertenecen a la familia Orthomyxoviridae. Existen cuatro tipos de virus Influenza A, B, C y D.
Los virus de tipo B y C son exclusivamente de humanos, mientras que D se ha aislado en animales.
Los virus tipo A y B tienen genomas segmentados de 8 segmentos, mientras que los C y D presentan 7 segmentos.
Aunque los virus de Influenza A y los virus de Influenza B son muy similares en estructura, los virus de Influenza B tienen una tasa de mutación más lenta.
Los virus tipo A puede infectar a humanos y a otros mamíferos, los principales reservorios de este tipo de virus son las aves migratorias, dentro de los virus de Influenza A se encuentran los virus de Influenza Aviar.
La clasificación de los subtipos de virus Influenza A, depende de la combinación de Hemaglutinina (HA) y Neuraminidasa (NA) que presenten, la diferencia de nucleótidos entre subtipos debe ser al menos un 30%. Hasta el momento se han reportado 18 subtipos de HA y 11 subtipos de NA.
El virus Influenza A es pleomórfico, su diámetro varía de 50 a 120 nm, cuenta con una bicapa lipídica tomada de la membrana celular.
Los virus Influenza A tienen un genoma de RNA de cadena sencilla, en sentido negativo, de 8 segmentos. Algunos segmentos del genoma de Influenza codifican para más de una proteína dependiendo del marco de lectura.
El genoma se encuentra empaquetado en estructuras conocidas como ribonucleoproteínas (RNP) conformadas por el RNA viral, la NP y el complejo de las polimerasas: Polimerasa básica 1 (PB1), polimerasa básica 2 (PB2) y polimerasa ácida (PA).
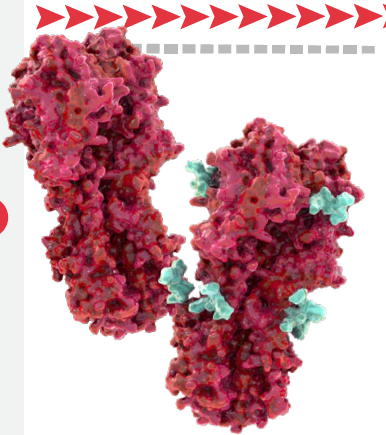
La HA es una glicoproteína integral de membrana, que se encuentra como principal antígeno de superficie, constituye alrededor del 80% de las proteínas expresadas en la superficie viral.
Se empaqueta en el retículo endoplásmico en forma de homotrímeros cilíndricos:
Los monómeros de HA son sintetizados como HA0, este precursor es inactivo, por lo que no puede infectar hasta ser cortado por enzimas proteolíticas.
En los virus aviares el sitio de corte determina la patogenicidad de la cepa viral, la cual puede ser de baja o alta patogenicidad.
Los virus de baja patogenicidad son digeridos por enzimas proteolíticas tipo tripsina (serina proteasa y triptasa clara), que se encuentran en el tracto respiratorio y en el tracto gastrointestinal, lo que conlleva a que la replicación viral se vea limitada.
Mientras que los virus de alta patogenicidad contienen un sitio de corte polibásico, con una mayor cantidad de argininas y lisinas, lo que permite que sean digeridos por enzimas intracelulares como las proteasas ubicuas, resultando en una diseminación viral sistémica.
La patogenicidad debe relacionarse con el comportamiento clínico, ya que los virus de alta patogenicidad deben ocasionar alta mortalidad al inocularse por vía intravenosa en pollos.
Los subtipos H5 y H7 han sido los únicos identificados como virus de alta patogenicidad, aunque hay variantes de estos mismos subtipos con un comportamiento y un sitio de corte de baja patogenicidad.
Los virus de alta patogenicidad ocasionan pérdidas importantes en las producciones avícolas, ya que pueden llegar a ocasionar la muerte del 100% de las aves afectadas.

Cambios en la Antigenicidad
La capacidad que tienen los virus de Influenza de cambiar su genoma para evadir la respuesta inmune del huésped se conoce como variación antigénica.
El proceso puede ser puntual y gradual, en ese caso la capacidad mutagénica conduce a cambios en las principales proteínas de superficie del virus, esto se conoce como deriva antigénica o antigenic drift.
Sin embargo, este no es el único mecanismo de cambio presente en los virus de Influenza.
Ya que estos virus tienen la capacidad de recombinarse cuando dos o más virus distintos infectan una misma célula, a esta capacidad de recombinación que conlleva al surgimiento de nuevos subtipos, que pueden modificar la capacidad de infectar nuevos huéspedes y saltar la barrera interespecie.
Lo cual se relaciona con el surgimiento de virus pandémicos, ya que pueden generarse virus contra los cuales el huésped no tenía ningún tipo de protección, a esto se le llama cambio antigénico o antigenic shift.
Desde la primera identificación de un virus de Influenza Aviar a la fecha, hemos detectado una alta capacidad de cambio y adaptación, tanto en presentaciones de subtipos, recombinaciones genéticas, mutaciones para evasión de la inmunidad.
O para cambiar la presentación clínica de la enfermedad, siendo la HA la proteína que más cambios presenta, es importante seguir de cerca los cambios por venir ya que al monitorear podremos estar listos para prevenir complicaciones asociadas a la alta tasa de mutación de los virus de Influenza Aviar.