Salud Animal
Next Generation Sequencing en avicultura, una innovadora herramienta de diagnóstico avícola
Para leer más contenidos de aviNews España Diciembre 2020
Salud Animal
Para leer más contenidos de aviNews España Diciembre 2020
Esta nueva herramienta diagnóstica permite la lectura de millones de secuencias génicas de forma masiva y simultánea en muy poco tiempo; facilitando y mejorando la identificación, clasificación y el diagnóstico de enfermedades.
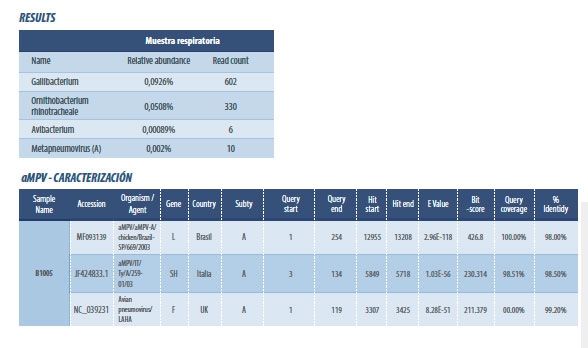
La tecnología NGS permite identificar, en una muestra de tejido (por ejemplo, una tarjeta FTA impregnada por una torunda del epitelio respiratorio o cloacal), todos los posibles organismos conocidos patógenos o no, que pudieran estar presentes.
El objetivo es realizar un informe completo que facilite la toma de decisiones sobre las medidas de bioseguridad a realizar (equipos de vacunación o recogida), calendarios de vacunación, diferentes opciones vacunales, etc.
Técnicas diagnósticas de biología molecular actuales

1. PCR en tiempo real (q-PCR)
PCR en tiempo real está indicado para detectar ADN.
El término PCR con transcripción reversa (RT-PCR) no debe confundirse con la PCR en tiempo real, también denominada PCR cuantitativa (q-PCR).
PCR en tiempo real se basa en la metodología del PCR convencional. La diferencia es que en:
Las PCR en tiempo real tienen las ventajas frente al PCR convencional de que son más sensibles, y lo más importante, además de indicarte si en una muestra hay presencia o no de un ADN específico de una determinada enfermedad, en caso de haya presencia, el resultado será semicuantitativo, dándonos una idea del grado de carga viral en esa muestra.
En el PCR en tiempo real, cada PCR es específico de una secuencia conocida concreta; por lo que tenemos que sospechar de una patología concreta para solicitar el PCR correspondiente.
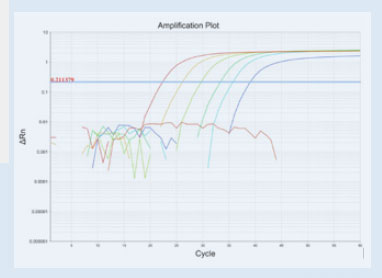
Ejemplo de resultados del PCR en tiempo real de 7 muestras
Las muestras representadas de color rojo, amarillo, verde claro, verde oscuro azul claro y azul oscuro, son positivas (en orden decreciente de carga viral). La muestra de color naranja es negativa.
2. Secuenciación
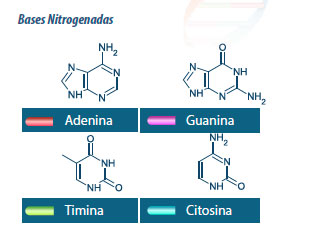
La secuenciación consiste en determinar el orden exacto de bases nitrogenadas de los nucleótidos que forman un fragmento de ADN o ARN de una muestra.
Normalmente, en laboratorios de diagnóstico se suele secuenciar una pequeña porción del genoma del patógeno, suficiente para clasificarlo en base a las similitudes genéticas con los otros genotipos.
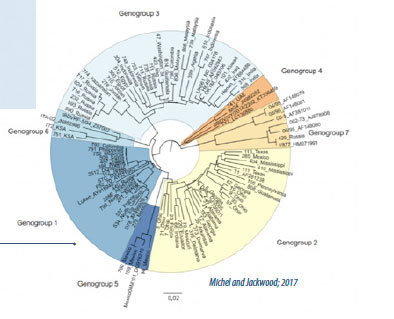
Árbol filogenético de los virus de Gumboro por comparación de un fragmento de 579 pares de bases de la región hipervariable del gen VP2.
La secuenciación se realiza mediante una modificación del PCR clásico, llamada método Sanger. Se utilizan los 2 cebadores, la polimerasa y los 4 desoxirribonucleótidos-trifosfato (sustrato del nuevo ADN), igual que en el PCR clásico. La diferencia es que se añaden también 4 “nucleótidos modificados” que tienen 2 diferencias con los “nucleótidos naturales”:
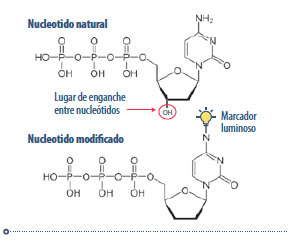
El resultado son multitud de fragmentos de ADN, que son copias parciales de diferente longitud (con una base de diferencia) de la secuencia modelo original. Todas estas copias parciales terminan con un nucleótido modificado (que contiene el marcador que refleja color).
Posteriormente, los fragmentos de ADN marcados fluorescentemente (con los 4 colores), se separarán por tamaño mediante electroforesis capilar. La detección automatizada de estos colores, proporciona una secuencia ordenada de los fragmentos en cromatogramas, que nos indica la secuencia génica de la muestra del ADN original (a una velocidad de 0,001 gigabases/día).
La nueva técnica de biología molecular “Next Generation Sequencing”
El termino NGS en realidad engloba varias tecnologías que producen la secuenciación masiva. La tecnología NGS más utilizada es la llamada secuenciación por síntesis.
Secuenciación por síntesis
En la fase previa a la secuenciación, se produce una desnaturalización (separación de la doble hélice) del ADN de la muestra (procedente del hisopo). Posteriormente estas cadenas monocatenarias se fragmentan en segmentos de menor tamaño.
La secuenciación tiene lugar sobre una placa de vidrio, que tiene prefijados unos “oligonucleótidos complementarios”, que son pequeños fragmentos de ADN que van a retener por un extremo, las porciones de ADN de la muestra. Así, cada fragmento de ADN monocatenario de la muestra, se unirá por uno de sus extremos a los diferentes “oligonucleótidos complementarios” fijados en la placa.
Posteriormente, la acción de la polimerasa, que utiliza estos “oligonucleótidos complementarios” como cebadores, sintetiza las cadenas de ADN complementarias (a las cadenas del ADN monocatenario de la muestra). Seguidamente, se suceden varios ciclos de elongación/ desnaturalización, se eliminan las secuencias complementarias y quedan únicamente una multitud de agrupaciones de secuencias idénticas de ADN monocatenario (llamadas “clusters”), iguales al ADN de la muestra, que están inmovilizadas sobre una superficie de vidrio.
En una fase posterior, se añaden, como en el método Sanger, “nucleótidos modificados” con marcadores luminosos y que tampoco permiten añadir más nucleótidos a partir de ellos, pero a diferencia del método Sanger, estos nucleótidos una vez incorporados a la cadena complementaria, dejan de ser “nucleótidos modificados”.
El proceso es el siguiente: tras la incorporación a la cadena complementaria de los nucleótidos modificados, se enjuaga la placa de vidrio para retirar los nucleótidos libres, quedando sólo los nucleótidos modificados que se han incorporado a la cadena complementaria de la muestra.
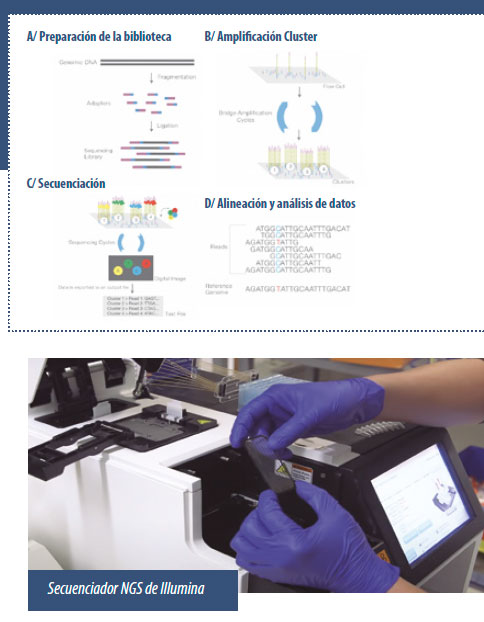
Finalmente, se “desmodifica” el nucleótido agregado ya leído, extrayendo del nucleótido modificado, el marcador luminoso y grupo bloqueador (añadiendo un grupo hidroxilo). Es decir, el nucleótido modificado una vez unido a la cadena, se “desmodifica”, convirtiéndose en normal.
Para conocer una nueva base del ADN de la muestra, se continua con un nuevo ciclo, que unirá un nuevo nucleótido modificado (que reflejará el color correspondiente) sobre el anterior nucleótido “desmodificado”, y así nucleótido a nucleótido.
La velocidad de lectura de esta técnica es de 4.000 gigabases/día (como referencia, el genoma humano tiene 3,2 gigabases).
La fase final es el análisis de los datos. La NGS genera millones de lecturas, por lo que se necesita un potente sistema informático que superponga estas lecturas, para formar las secuencias génicas parciales de los diferentes organismos presentes en la muestra; Y así poderlos comparar con las secuencias génicas de los organismos conocidos hasta la fecha, patógenos o no.
Conclusión
La característica más destacada de la NGS es el poder de descubrimiento. El NGS es un enfoque libre de hipótesis que no requiere un conocimiento previo a priori del genoma, es decir no requiere la sospecha de una patología ni petición especifica.
Secuencia a mayor profundidad, ya que se puede secuenciar amplias porciones del genoma del patógeno, por lo que proporciona un mayor poder de descubrimiento para detectar genes nuevos y una mayor sensibilidad para identificar nuevas variantes.
Requiere menos ADN / ARN en la muestra que la PCR en tiempo real (cantidades de nanogramos ya son suficientes).